版權歸原作者所有,如有侵權,請聯係我們(men)
1806 年,23 歲的德國藥劑師 Sertürner 從(cong) 罌粟中首次分離出單體(ti) 嗎啡,現代天然藥物化學研究自此起步。在此基礎上,德國化學家 Friedrich Wǒhler 在 1828 年成功實現了尿素的人工合成,這也標誌著有機化學學科的正式誕生。可以說,正是人類對生物活性天然產(chan) 物 (natural product, NPs) 的持續研究促成了有機化學學科的建立。
所謂生物活性天然產(chan) 物 (NPs) ,其實是自然界長期進化的物質實體(ti) ,是生物活性物質和實用藥物研發的重要源泉。在藥物研發進程中,NPs 對於(yu) 癌症和傳(chuan) 染病治療藥物的創新有著巨大貢獻。但時至今日,NPs 仍在篩選、分離、表征、優(you) 化等各方麵存在技術障礙。其中,從(cong) 複雜混合物中分離 NPs 可謂是最為(wei) 嚴(yan) 峻的挑戰之一,這也成為(wei) 了藥物研究的一大瓶頸。
為(wei) 了解決(jue) 這一瓶頸, 中南大學湘雅醫院藥學部劉韶教授團隊,創新性地建立了一種可全麵挖掘天然藥物藥效成分的整合分子網絡框架 (integrated molecular networking workflow for NP dereplication, IMN4NPD),不僅(jin) 加快了分子網絡中廣泛集群的去複製 (dereplication),而且對現有研究方法中經常被忽略的自循環與(yu) 成對節點提供標注。相關(guan) 研究成果日前被發表於(yu) 美國化學會(hui) (ACS) 期刊 Analytical Chemistry。

論文地址:
https://doi.org/10.1021/acs.analchem.3c04746
IMN4NPD:集成多種計算工具,由光譜相似度驅動的分子網絡
IMN4NPD 的核心工作原理是由光譜相似度驅動的分子網絡。它通過集成並協同 NPClassifier, molDiscovery 和 t-SNE 網絡等多種計算工具,從(cong) 而幫助研究人員快速識別特定類別的化合物,同時還能簡化分子網絡節點中的標注。
* NPClassifier:一種基於(yu) 深度神經網絡的天然產(chan) 物結構分類工具
* molDiscovery:一種質譜數據庫搜索方法
一般而言,IMN4NPD 的工作流程可分為(wei) 3 步:
第一步,對原始 LC-MS 數據進行預處理,以生成分子網絡或基於(yu) 特征的分子網絡。隨後,基於(yu) 深度神經網絡的 NP 分類工具 SIRIUS,通過 NPClassifier 對複合類進行係統分類。
第二步,該研究通過 GNPS (Global Natural Product Social Molecular Networking),進行了基於(yu) MS/MS 光譜數據庫的去複製實驗,然後通過 molDiscovery 進行基於(yu) 計算機數據庫的去複製。
第三步,研究人員利用 MS/MS 光譜特征的相似度生成 t-SNE 網絡,並對每個(ge) 節點的化合物進行化學分類,以精確定位、並去複製分布在自循環網絡中的特定化合物類別。
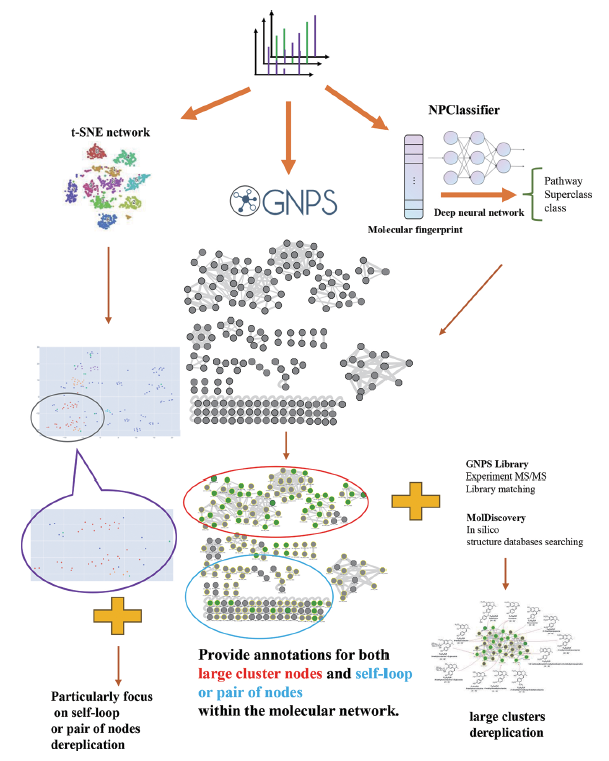
IMN4NPD 工作流程示意圖
可用性評估:探索異喹啉類似物,在分子網絡中迅速識別特定化合物簇
為(wei) 了評估 IMN4NPD 工作流程的性能和優(you) 勢,該研究重新分析了蓮子心的乙醇提取物。蓮子心是蓮蓬中的胚芽部分,是一種富含雙苄基異喹啉、單苄基異喹啉和阿樸啡等多種生物堿的中藥植物,可用於(yu) 治療失眠、遺精、心率失調、高血壓等症狀。
基於(yu) 實驗性 MS/MS 光譜數據庫,該研究最初對分子網絡中的單個(ge) 節點進行化學分類,從(cong) 而在分子網絡中迅速識別出特定的化合物簇,以探索新的異喹啉類似物。在查看了分子網絡中每個(ge) 特征映射的化學分類結果後,研究人員發現,很容易就能找到與(yu) 異喹啉類似物相對應的某些化合物簇,同時,異喹啉類化合物主要分布在分子網絡中的四個(ge) 簇中。
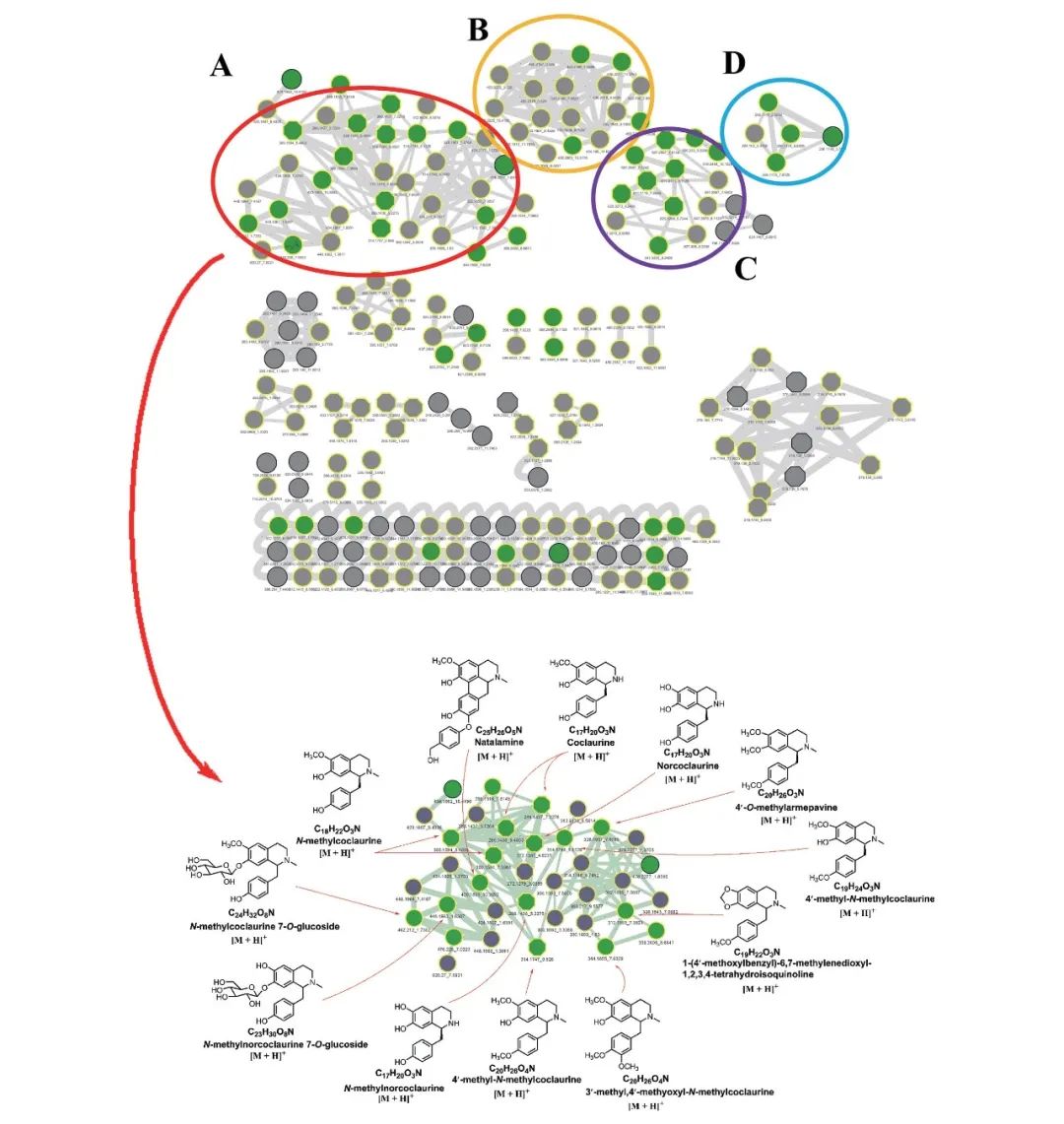 異喹啉類化合物分布圖
異喹啉類化合物分布圖
該研究還發現,通過實驗性 MS/MS 光譜數據庫(如 GNPS 數據庫)隻能成功地去複製大型簇中數量有限的特征。因此,該研究采用了最先進的矽學片段算法 molDiscovery 進行結構數據庫匹配。這種基於(yu) 實驗和矽學 MS/MS 圖譜數據庫的去複製方法,增強了在分子網絡中、尤其是在大型集群中,及時、方便地標注物質結構的能力。
以單苄基異喹啉生物堿中的集群 A 為(wei) 例,該集群由 36 個(ge) 節點組成,其中 MS 數據庫隻標注了 7 個(ge) 節點,Structure 數據庫標注了 35 個(ge) 節點,MS 和 Structure 數據庫同時標注了 8 個(ge) 節點。值得注意的是,這其中有一個(ge) m/z 344.1855 節點 (tR=7.6329) 被 MS 結構數據庫完全標注,這表明候選結構為(wei) 3′-O-methyl-4′-methoxy-N-methylcoclaurine(如上圖所示)。
通過進一步分析,該節點連續損失了 NH3CH3、CH3OH 和 H2O,隨後發生了環裂解、α 裂解和 β 裂解,分別在 m/z 107.0496、137.0597、151.0757、175.0750、205.1098、235.0752、267.1017、299.1271 和 312.1590 處產(chan) 生碎片離子。
經 Structure 數據庫鑒定,m/z 448.1963(tR = 1.6287)的結點為(wei) N-methylnorcoclaurine 7-O-glucoside。另一個(ge) m/z 312.1593 (tR = 7.3621) 節點則顯示了包含 1 個(ge) 單苄基異喹啉在內(nei) 的四個(ge) 候選結構。與(yu) m/z 344.1855 節點 (tR=7.6329) 相比,該節點在 m/z 190.0862 (C11H12NO2) 處存在碎片離子,表明這是一個(ge) 亞(ya) 甲基二氧基。
研究結果:基於(yu) 深度神經網絡,從(cong) t-SNE 網絡的角度對比三大研究算法
與(yu) MolNetEnhancer 相比,IMN4NPD 采用基於(yu) 深度神經網絡的 NP 分類工具 NPClassifier,來單獨分類分子網絡中的每個(ge) 特征,而不是整個(ge) 簇或分子家族。該研究使用了改進過的餘(yu) 弦相似度計算相似矩陣,並以此生成 t-SNE 網絡。同時,該研究還通過 NPClassifier 基於(yu) 每個(ge) 節點的 MS/MS 光譜數據對其進行分類,並將這些分類映射到 t-SNE 網絡中。
在傳(chuan) 統的分子網絡觀點中,異喹啉一般由三個(ge) 大簇 (簇 A-C) 和一個(ge) 小簇 (簇 D) 共同組成。從(cong) t-SNE 網絡的角度來看,很明顯,異喹啉的四個(ge) 集群節點被緊密分組,形成了不同的集群區域。但值得注意的是,從(cong) t-SNE 網絡的角度來看,分子網絡中的簇 A 可以進一步分為(wei) 兩(liang) 個(ge) 更小的簇。此外,t-SNE 可以有效定位異喹啉類節點,從(cong) 而大大減輕了相關(guan) 節點的結構解析工作。
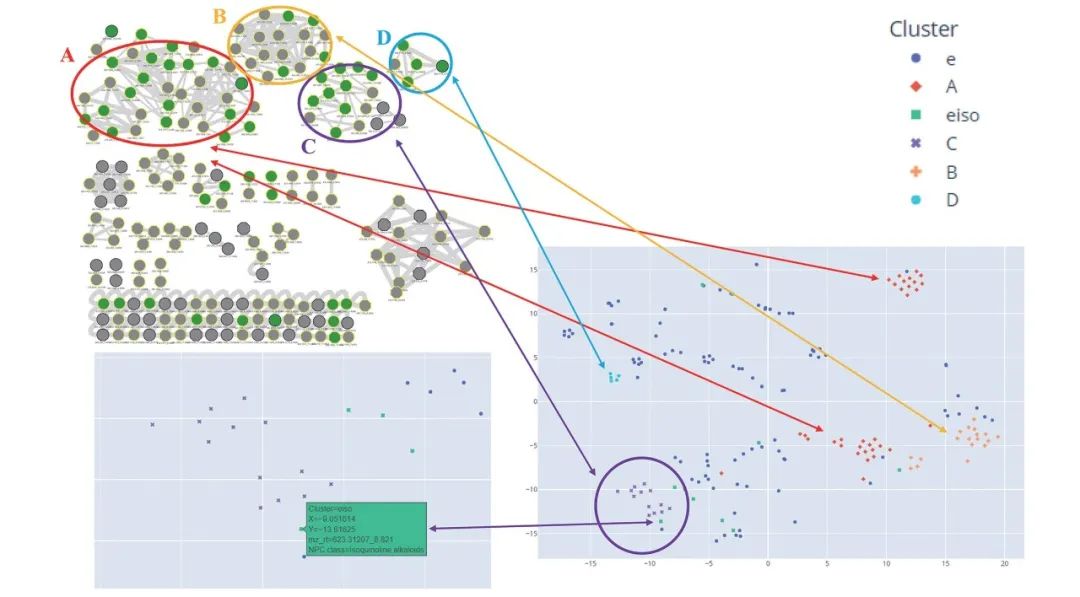 異喹啉在 t-SNE 圖譜中的四個(ge) 聚類區域
異喹啉在 t-SNE 圖譜中的四個(ge) 聚類區域
修正餘(yu) 弦相似度方法麵對多種學修飾的化合物光譜存在局限性,該研究還選擇了 Spec2Vec 和 MS2DeepScore 等相似度算法,並以此生成 t-SNE 網絡。基於(yu) Spec2Vec,異喹啉依然在分子網絡中形成四大簇區。
但基於(yu) MS2DeepScore,異喹啉的大簇 A 和 B 的節點間隔很近,形成了幾個(ge) 聚類區域,但大簇 C 中的節點分散在了整張圖中,這為(wei) 後續分析帶來了挑戰。
 多種光譜相似度算法生成的 t-SNE 圖譜比較
多種光譜相似度算法生成的 t-SNE 圖譜比較
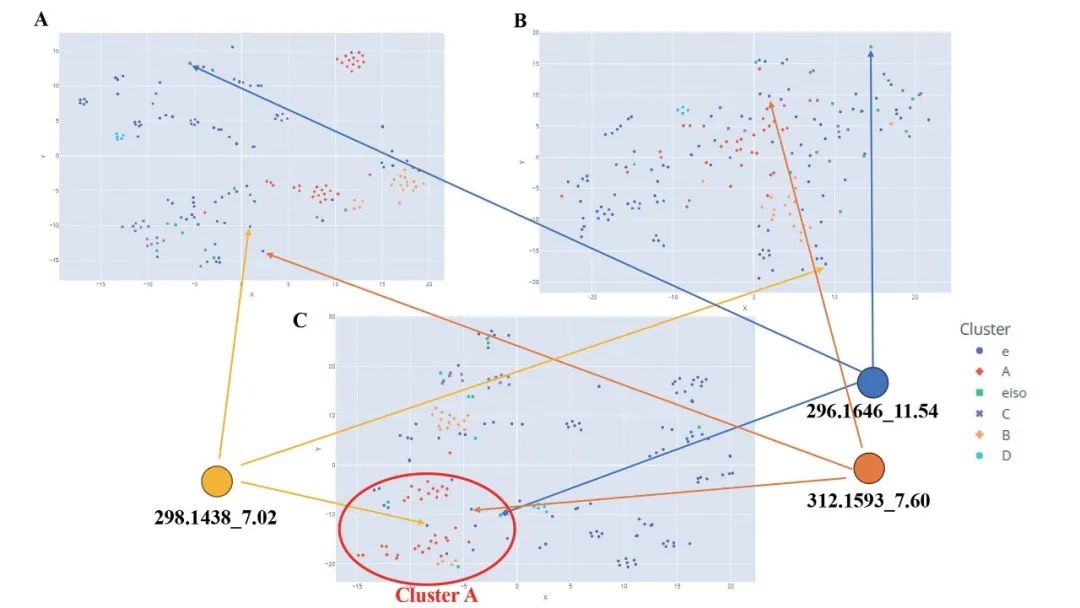
一個(ge) 有趣的現象是,m/z 296.1646節點 (tR = 11.54) 在修正餘(yu) 弦相似度和 MS2DeepScore 相似度的 t-SNE 圖中,均遠離異喹啉相關(guan) 的節點聚類區域,但在基於(yu) Spec2Vec 光譜相似度的 t-SNE 圖中,該節點與(yu) 大簇 A 的聚類區域相鄰。這類自換節點可能代表了一類異喹啉化合物,在進一步比較後可確認該節點是阿樸啡類生物堿。
因此,化合物化學分類和 t-SNE 網絡,可分別提供關(guan) 於(yu) 特征的不同信息,一定程度上減少了假陰性的出現。
此外,基於(yu) Spec2Vec 光譜相似度的 t-SNE 網絡,大簇 A 附近存在 m/z 298.1438 (tR = 7.02) 和 m/z 298.1438 (tR = 7.60) 兩(liang) 個(ge) 節點,這兩(liang) 個(ge) 節點是分子網絡中的自換節點和對節點。盡管沒有被歸類為(wei) 異喹啉化合物,但它們(men) 與(yu) 異喹啉大簇 A 結構相似。進一步分析可知,m/z 298.1438 (tR = 7.02) 是一種已知的阿樸啡類生物堿——nornuciferidine,m/z 298.1438 (tR = 7.60) 也顯示出與(yu) nuciferine 和 nornuciferidine 相類似的阿樸啡類生物堿。
通過對以上三個(ge) 節點的研究發現,它們(men) 都屬於(yu) 阿樸啡類生物堿,這與(yu) 單苄基異喹啉類生物堿不同。在利用修正餘(yu) 弦相似度和 MS2DeepScore 相似度時,這三個(ge) 節點遠離單苄基異喹啉類生物堿相關(guan) 節點的聚類區域大簇 A,但基於(yu) Spec2Vec,這三個(ge) 節點卻可在大簇 A 附近被發現。
這種差異表明 Spec2Vec 光譜相似性在準確捕捉異喹啉類化合物相似結構方麵的卓越能力。
人工智能在天然產(chan) 物研究中的應用加速
近年來,受益於(yu) 各種現代技術的迅猛發展,在天然生物活性分子的研究中湧現出了一大批基於(yu) LC-MS/MS 和 NMR 技術,並集成生物信息學、代謝組學、計算機科學等多學科技術手段的新策略和新方法。尤其是,隨著人工智能和機器學習(xi) 算法開始融入天然產(chan) 物研究工作,進一步為(wei) 研究人員帶來了新一輪的生產(chan) 力革命。
最初,人工智能的應用集中在有機分子的數字化,以及使用降維技術繪製 NP 化學空間圖。後來,研究者通過開發機器學習(xi) 二元分類器來預測 NP 的生物功能。如今,神經網絡架構開始被用於(yu) 基因組挖掘和分子設計,深度學習(xi) 算法在藥物發現和分子信息學領域越來越受歡迎。
所以,我們(men) 可以看到,產(chan) 學研各界近年來均加快了相關(guan) 研究的步伐。2022 年,國家超級計算廣州中心就聯合中山大學、星藥科技、美國麻省理工學院和佐治亞(ya) 理工學院,基於(yu) 「天河二號」的強大計算和存儲(chu) 能力,提出了一種深度學習(xi) 驅動的生物逆合成路徑導航工具 BioNavi-NP。
而在企業(ye) 界,天然產(chan) 物的研究也在不斷加速。2023 年,天士力醫藥集團與(yu) 華為(wei) 雲(yun) 達成合作,雙方將結合天然產(chan) 物現代化研究數據等,共建中醫藥領域垂直大模型。
然而,天然產(chan) 物數據庫仍然是科研進程中的一大挑戰。當前,全世界主流的天然產(chan) 物數據存儲(chu) 庫,包括生物合成基因簇的最小信息 (MIBiG)、天然產(chan) 物圖譜 (NP 圖譜)、全球天然產(chan) 物分子網絡 (GNPS)、天然產(chan) 品磁共振數據庫 (NP-MRD) 等,但這些數據庫的覆蓋率較低,並且存在較為(wei) 常見的數據錯誤問題,這些都阻礙了人工智能在天然產(chan) 物藥物發現方麵的進展。
近年來,中國科學家屠呦呦、日本科學家大村智和愛爾蘭(lan) 科學家 William C. Campbell 等多位研究者因在天然產(chan) 物全合成方麵的成就獲得了諾貝爾化學獎提名。毫無疑問,隨著天然產(chan) 物的重要性不斷凸顯,人工智能在天然產(chan) 物研究方麵的融合也即將按下加速鍵。
歡迎掃碼關(guan) 注深i科普!
我們(men) 將定期推出
公益、免費、優(you) 惠的科普活動和科普好物!



- 參加最新科普活動
- 認識科普小朋友
- 成為科學小記者
 會員登錄
會員登錄














 深圳市華強北街道 華新地鐵站A1出口24小時科學銀行
深圳市華強北街道 華新地鐵站A1出口24小時科學銀行












